ÍNDICE
|
12. Ciclos biológicos 13. Posibles modelos de replicación del ADN 14. El experimento de Meselson y Stahl 15. Otras característcas de la replicación 16. La replicación del ADN 1. Fase de iniciación 2. Fase de elongación 3. Fase de terminación 17. Replicación del ADN en eucariotas 18. Ideas fundamentales 19. Repaso 20. Tareas 21. Prácticas 22. Vídeos 23. Otras presentaciones 24. Cuestiones |
1. Conocimientos previos
2. ESQUEMAS
3. PRESENTACIONES

















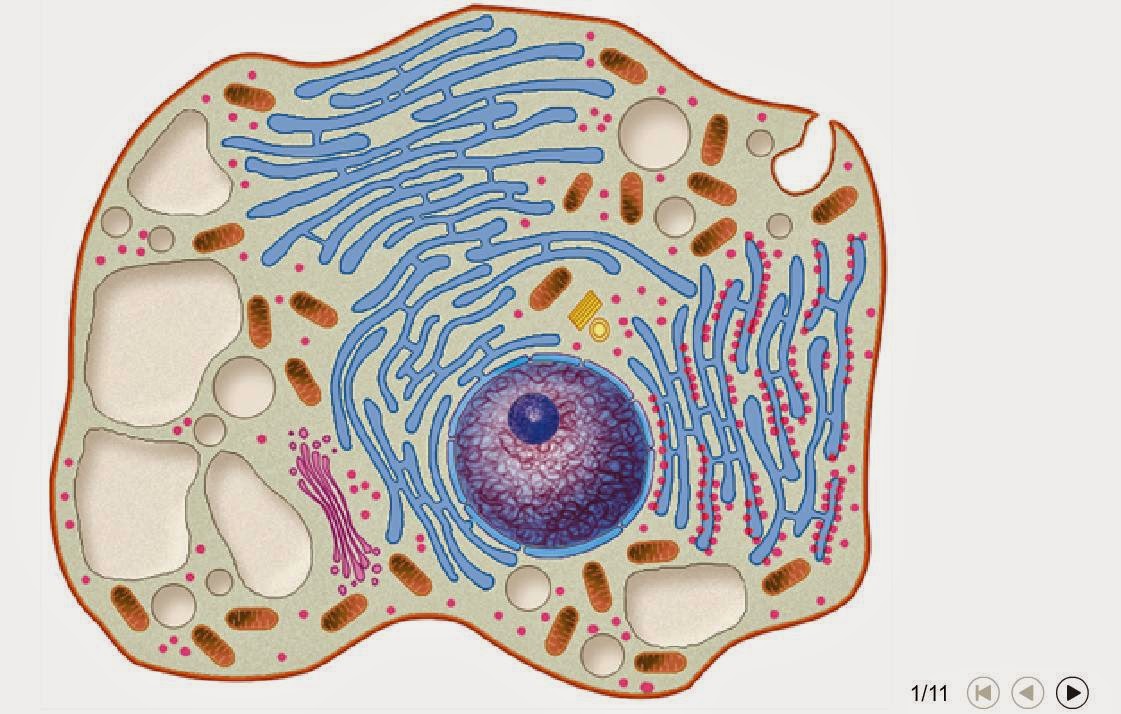
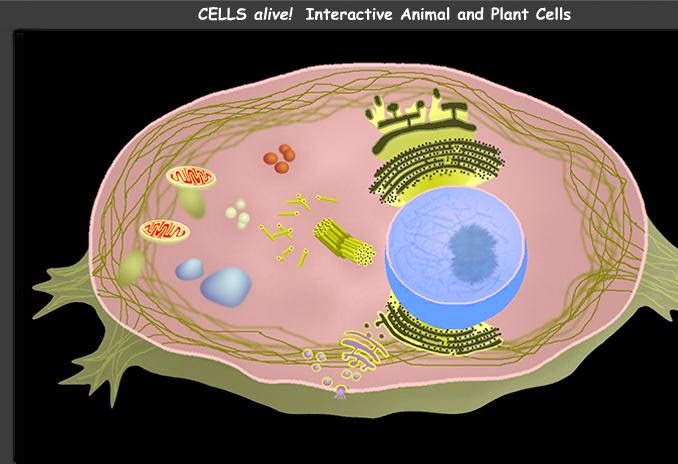
Imágenes del núcleo
5. EL NÚCLEO CELULAR
El núcleo es la estructura más destacada de la célula eucarionte, tanto por su morfología como por sus funciones. Su tamaño es variable (5 a 10 mm) al igual que su ubicación siendo en la mayoría de los tipos celulares central.
El núcleo tiene tres funciones primarias, todas ellas relacionadas con su contenido de ADN. Ellas son:
- Almacenar la información genética en el ADN.
- Recuperar la información almacenada en el ADN en la forma de ARN.
- Ejecutar, dirigir y regular las actividades citoplasmáticas, a través del producto de la expresión de los genes: las proteínas.
En el núcleo se localizan los procesos a través de lo cuales se llevan a cabo dichas funciones. Estos procesos son:
- La duplicación del ADN y su ensamblado con proteínas (histonas) para formar la cromatina.
- La transcripción de los genes a ARN y el procesamiento de éstos a sus formas maduras, muchas de las cuales son transportadas al citoplasma para su traducción.
- La regulación de la expresión genética.



5.1. Estructura del núcleo
El núcleo está rodeado por la envoltura nuclear, una doble membrana interrumpida por numerosos poros nucleares. Los poros actúan como una compuerta selectiva a través de la cual ciertas proteínas ingresan desde el citoplasma, como también permiten la salida de los distintos ARN y sus proteínas asociadas.
La envoltura nuclear es sostenida desde el exterior por una red de filamentos intermedios dependientes del citoesqueleto, mientras que la lámina nuclear, la cual se localiza adyacente a la superficie interna de la envoltura nuclear, provee soporte interno.
El núcleo también tiene un nucleoplasma, en el cual están disueltos sus solutos y un esqueleto filamentoso, la matriz nuclear la cual provee soporte a los cromosomas y a los grandes complejos proteicos que intervienen en la replicación y transcripción del ADN.
Los cromosomas aparecen ocupando lugares específicos. Los genes que codifican productos relacionados, aunque estén localizados en diferentes cromosomas, pueden estar ubicados próximos en el núcleo interfásico. Por ejemplo, los cromosomas humanos 13, 14, 15, 21 y 22 poseen un gran número de genes que codifican para ARNr. Dichos cromosomas están agrupados de tal forma que los genes de los ARNr están todos juntos y confinados en el nucléolo, el lugar donde se sintetizan, procesan y ensamblan los ARNr. Esta separación física asegura que los ARNr puedan ser eficientemente ensamblados dentro de las subunidades ribosomales.
En el núcleo, los genes transcripcionalmente activos tienden a estar separados de los inactivos. Los activos se encuentran ubicados centralmente, mientras que los silentes están confinados próximos a la envoltura nuclear.
Tan pronto como las células entran en mitosis o meiosis, los fragmentos de la matriz nuclear dirigen la condensación de los cromosomas, constituyéndose en la parte central de los mismos.




La envoltura está formada por dos membranas concéntricas interrumpidas por poros nucleares y por la lámina nuclear.

Las membranas delimitan un espacio de 10 a 50 nm, el espacio o cisterna perinuclear. La membrana externa en contacto con el citoplasma tiene ribosomas adheridos, que sintetizan las proteínas que se vuelcan al espacio perinuclear. El espacio perinuclear se continua con el REG.
La membrana interna posee proteínas integrales que le son propias, que se unen a la lámina nuclear y a los cromosomas.
La lámina nuclear, capa fibrosa de 10 a 15 nm en la que apoya la membrana interna, está formada por proteínas del tipo de los filamentos intermedios, polímeros de lamina o laminina nuclear. Ellas se unen a las proteínas integrales de membrana.
La fosforilación de las laminas provoca el desensamble de la lámina nuclear causando la ruptura de la envoltura al iniciode la división celular.
La lámina nuclear confiere estabilidad mecánica a la envoltura nuclear. Además, al interactuar con la cromatina participa en la determinación de la organización tridimensional del núcleo interfásico.
Si bien la formación de la lámina no se requiere durante los pasos iniciales, la organización de la envoltura es indispensable para el crecimiento posterior y el mantenimiento de su integridad. Las laminas se incorporan luego que la cisterna perinuclear rodea al ADN y se inicia el transporte entre el núcleo y el citoplasma.




La envoltura nuclear es un derivado del sistema de endomembranas, siendo esto evidente al inicio de la división celular, cuando la envoltura se desorganiza y pasa a formar parte del sistema de cisternas y vesículas del retículo endoplásmico.
La aparición de la envoltura nuclear permitió que los eucariontes aislaran los procesos genéticos principales, como la autoduplicación del ADN o la síntesis de ARN. Además esto posibilitó que el ARNm se modifique dentro del núcleo antes de ser traducido en los ribosomas. Estas modificaciones no ocurren en los procariontes, ya que a medida que la ARN polimerasa sintetiza el ARN, simultáneamente el extremo 5’ se une al ribosoma y comienza la traducción.
La envoltura nuclear presenta estructuras discoidales llamadas complejos de poro nuclear (CPN).
El número de CPN es variable, incrementándose a medida que aumenta la actividad celular. En una célula de mamífero hay entre 3000 a 4000 complejos de poro. Cada CPN es una estructura macromolecular compleja constituida por un gran número de proteínas de disposición octamérica. Está formado por:
- Ocho columnas proteicas, que forman las paredes laterales del poro.
- Un anillo externo, formado por ocho unidades proteicas.
- Un anillo interno, también con estructura octamérica.
- Proteínas de anclaje que fijan cada columna al espacio perinuclear.
- Proteínas radiales que se proyectan desde las columnas hacia la luz del poro, a manera de diafragma
- Proteínas fibrilares fijas al anillo interno y externo. En la cara nuclear convergen para formar una canastilla o cesta. A lo largo de estas fibrillas se ubicannucleoporinas que intervienen en el transporte de sustancias a través del poro.
- Un poro central o abertura.


La luz de los CPN suele presentarse obturada por las proteínas que circulan a través del poro, como por las carioportinas (Kap) que actúan como eficientes transportadores en el tráfico núcleo/citoplasma.
Los CPN presentan uno o varios canales acuosos a través de los cuales las pequeñas moléculas solubles en agua difunden (transporte no regulado). Las moléculas de mayor peso molecular son transportadas en forma activa, por lo que requieren energía y moléculas transportadoras.
Se importan dentro del núcleo:
- Las proteínas sintetizadas en el citoplasma necesarias para ensamblar los ribosomas.
- Los factores de transcripción requeridos en la activación o inactivación de los genes.
- Los factores de empalme necesarios en el proceso de maduración de los ribosomas.
- Las moléculas y macromoléculas ensambladas y exportadas desde el núcleo al citoplasma incluyen:
- Las subunidades ribosomales
- ARNm
- ARN de transferencia
- Factores de transcripción que son devueltos al citoplasma para ser reutilizados.
Los mecanismos implicados en el transporte a través del poro son diferentes al transporte de proteínas en las membranas de otros organelos . Por ejemplo, las proteínas nucleares son transportadas a través del poro manteniendo su conformación plegada, por el contrario las proteínas que no se localizarán en el núcleo se despliegan durante el transporte.
Los complejos de poro nuclear hacen de la envoltura nuclear una barrera selectiva entre el núcleo y el citoplasma. Estos complejos constituyen la principal vía de comunicación entre el compartimiento nuclear y citoplasmático de la célula ante el pesado tráfico molecular. Aun cuando las proteínas pequeñas y otras moléculas viajan a través de los canales periféricos, las proteínas de gran tamaño deben poseer una etiqueta para ingresar por el canal central. Estas proteínas sintetizadas en el citoplasma contienen la señal de localización nuclear (nuclear signal localization, NSL).
Tampoco los ARN pueden salir de núcleo por sí mismos. Ellos salen a través del complejo de poro con una proteína especial que posee una señal nuclear de exportación (nuclear export signal, NES). Ambas NSL y NES consisten en una secuencia corta de aminoácidos, muchos de los cuales tienden a estar ubicados centralmente dentro de las proteína.
Las moléculas de mayor tamaño requieren de una proteína “transbordadora” o carioportina. La superfamilia de carioportinas esta integrada por: importinas . exportinas y transportinas .
Las importinas son heterodímeros, formados por dos subunidades, la subunidad-a se une a la NSL de la proteína nuclear permitiendo la unión con la subunidadad-b. esta unión origina una “importina funcional” que lleva unida a la proteína nuclear a ser transportada.
El complejo importina funcional se pone en contacto con los filamentos citosólicos, donde guiado por las nucleoporinas (Nup), llega al poro central. La translocación de complejo importina/carga es regulado por la pequeña RanGTPasa, que se une a la subunidad b de la importina. Esta b-importina es la encargada de interactuar con el poro provocando su dilatación y posibilitando el ingreso de la proteína nuclear. La translocación de proteínas es un proceso activo. Cuando el complejo penetra al interior del núcleo, las subunidades de importina se separan y la carga es liberada. La disociación de las subunidades causa entonces un nuevo cambio en su forma, dejando al descubierto la NES de cada subunidad. Otras proteínas en el poro central reconocen la NES y retornan las subunidades al citoplasma.




6.3. Exportación del ARN
Los ARN maduros se asocian a proteínas llamadas transportinas, las cuales actúan como transbordadores permitiendo el pasaje de ARN al citoplasma. En la fig. 10.6 se esquematiza como el ARNm es llevado fuera del núcleo. Los ARNm maduros que presentan la poli A se asocian con varias proteínas, formando una partícula de ribonucleoproteína (RNP). Estas partículas se mueven linealmente a través de la canasta nuclear. Al igual que las importinas, las RNP son recicladas hacia el núcleo. En el citoplasma, las CRBP ( del inglés, cytoplasmic RNA-binding proteins) reemplazan a las RNP para guiar a los ARNs a sus destinos citosólicos correctos.




El núcleo contiene los cromosomas de la célula. cada cromosoma consiste en una molécula única de ADN con una cantidad equivalente de proteínas. Colectivamente, el ADN con sus proteínas asociadas se denomina cromatina. La mayor parte de las proteínas de la cromatina consisten en copias múltiples de cinco clases de histonas.
Estas proteínas básicas son ricas en residuos de arginina y lisina cargados positivamente. Por esta razón se unen estrechamente con los grupos fosfatos (cargados negativamente) del ADN.
La cromatina también contiene pequeñas cantidades de una amplia variedad de proteínas no histónica. La mayoría de ellas son factores de transcripción (por ej., el receptor esteroide), siendo su asociación con el ADN pasajera. Estos factores regulan que parte del ADN será transcripta en ARN.




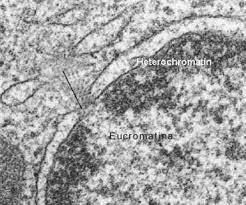
La observación a través del microscopio óptico de un núcleo interfásico, nos permite distinguir dos tipos de cromatina. La eucromatina o cromatina laxa, de localización central, y la heterocromatina o cromatina densa, en la periferia del núcleo (Fig.10.7). La heterocromatina representa aproximadamente el 10% del total de cromatina y es considerada transcripcionalmente inactiva
Si el núcleo celular se incuba con nucleasas, enzimas que digieren el ADN, las secuencias que primero se digieren son las que portan los genes expresados por la célula, lo que corrobora el menor grado de condensación de la eucromatina.




Cuando el cromosoma en interfase se esparce artificialmente sobre agua, tiene la apariencia de un collar de perlas. Las perlas son los nucleosomas, las unidades de enrollamiento de la cromatina.
Los nucleosomas están formados por un centro o "core" de histonas. Dicho centro posee dos copias de cada una de las siguientes histonas: H2A; H2B; H3 y H4



Alrededor de 60 pares de bases de ADN unen un nucleosoma con el próximo. Cada región de unión es el ADN espaciador. La quinta histona, la H1, conecta a los nucleosomas y actúa como una banda de goma, manteniéndolos juntos dentro de una misma cuerda enrollada. esta estructura se conoce como fibra de 10nm, siendo el primer grado del empaquetamiento de la cromatina.
Los nucleosomas se organizan, a su vez, en fibras de 30nm (solenoide), girando a manera de resorte alrededor de un eje virtual. Esta estructura es mantenida por la interacción de las H1 de nucleosomas cercanos.




En el siguiente nivel de empaquetamiento, las fibras de 30 nm se organizan en una serie de bucles o asas superenrolladas. Estos bucles se estabilizan gracias a la interacción con las proteínas de la matriz nuclear o andamiaje nuclear (“scaffold”).
Cada bucle de cromatina representa un dominio funcional o unidad de replicación (Fig. 10.10e). Estos dominios contienen alrededor de 100.000 pares de bases, extensión de ADN suficiente para acomodar varios genes de tamaño promedio. Algunos genes, sin embargo, pueden abarcar varios dominios adyacentes de un cromosoma. Cada cromosoma puede tener cien o más dominios.Durante la profase, los cromosomas aparecen en forma más condensada, alcanzando la cromatina su mayor nivel de condensación en metafase(Fig. 10.10f). La organización de los cromosomas envuelve la fosforilación de la H1 y otras proteínas, lo cual causa el plegamiento y empaquetamiento aún más compacto de la cromatina. El andamiaje o matriz nuclear se convierte en el centro de la estructura del cromosoma, y como la compactación continúa, éste se pliega modo de acordeón






El grado de condensación de los dominios de cromatina se mantiene principalmente debido a la asociación con la matriz nuclear y a proteínas asociadas como la topoisomerasa II o girasa, encargada de controlar el grado de superenrollamiento del ADN . La unión entre la cromatina y la matriz se da a nivel de zonas altamente conservadas, denominadas secuencias SAR o MAR (scaffold associated regions/ matrix attachment regions). Las SAR son regiones de varios cientos de pares de bases ricas en residuos de adenina y timina, abundantes en la heterocromatina.
Con coloraciones especiales los cromosomas, revelan diferencias estructurales de importancia funcional. Las bandas oscuras consisten en cromatina altamente condensada, mientras que las bandas claras se corresponden con cromatina más laxa.




El examen de la cromatina en bandas claras y oscuras revela que ambos tipos de cromatina están acomodados en bucles de distintos tamaños y que a su vez se proyectan desde el andamiaje plegado. El andamiaje está muy plegado en la heterocromatina, y es más lineal en las bandas de eucromatina, formando bucles más amplios. Las histonas de la eucromatina están fuertemente acetiladas. Estos cambios afectarían el grado de empaquetamiento de la eucromatina, haciéndola más accesible para la transcripción de sus genes.
Las características de la hetero y eucromatina son:
Tabla 10.1 - Características de la Cromatina
| ||||
Tipo de cromatina
|
Estadofísico
|
Cambio químico
|
Tipo de genes
|
Replicación
|
Eucromatina
|
Laxa
|
Acetilada
|
Activos
|
Fase S temprana
|
Heterocromatina
|
condensada
|
Metilada
|
Silentes
|
Fase S tardía
|
Los cromosoma en metafase también poseen un revestimiento de RNP. Dicho revestimiento deriva de los componentes del nucléolo. Estos cromosomas se constituyen en elvehículo para dividir el material nucleolar entre las futuras células hijas. El empaquetamiento de la cromatina permite confinar al ADN dentro del núcleo La molécula de ADN de un cromosoma humano contiene 50 x 106 pares de nucleótidos en el cromosoma más pequeño (1.7 cm con la molécula extendida) a 250 x 106 pares de nucleótidos en el más largo ( 8.5 cm). Midiendo extremo con extremo el total de cromosomas de una célula humana diploide, el ADN se extiende más de 2 metros. El empaquetamiento del ADN en forma de cromatina, no solamente le permite entrar dentro de los límites del núcleo, sino también lo protege del ataque de las nucleasas.
Cada cromosoma eucariota consiste en una molécula simple de ADN de alrededor de 150 millones de pares de nucleótidos.
La molécula de ADN en el cromosoma eucariota es lineal, por lo tanto posee dos extremos (en contraste con el cromosoma bacteriano que es circular).
La molécula de ADN de un cromosoma típico eucariota contiene:
- Un conjunto lineal de genes que codifican para ARN y proteínas interrumpido por
- Muchas secuencias de ADN no codificante.
El ADN no codificante incluye:
- Secuencias de aproximadamente 170 nucleótidos de ADN satélite, repetidas miles de veces, que corresponden al centrómero.
- Secuencias repetitivas en los extremos del cromosoma llamadas telómeros.
- Múltiples secuencias señalizadoras altamente conservadas, denominadas origen de replicación (ORI) , necesarias para que se realice la duplicación del ADN en un tiempo breve.


El centrómero es una constricción primaria localizada centralmente o hacia los extremos de cada cromosoma.
El ADN centromérico como ya mencionamos es altamente repetitivo y se encuentra siempre condensado siendo parte de la heterocromatina.
Los telómeros son cruciales en la vida de la célula. Ellos son necesarios para la duplicación completa del cromosoma, los protegen de las nucleasas, evitan que los extremos del cromosoma se fusionenentre sí y facilitan la interacción del cromosoma con la envoltura nuclear.
5 '... ..TTAGGG TTAGGG TTAGGG TTAGGG TTAGGG TTAGGG..... 3 '
3 '... ..AATCCC AATCCC AATCCC AATCCC A..... 5 '
La telomerasa es una ribonucleoproteína, la cual provee un molde de AAUCCC que guía la inserción de la secuencia TTAGGG. Entonces la telomerasa es una retrotranscriptasa, sintetiza ADN a partir de un molde de ARN.
Las células con telomerasa activa pueden compensar el acortamiento de los telómeros durante la duplicación del ADN.
La telomerasa activa se encuentra solamente en:
- Las células de la línea germinal, incluyendo células troncales embrionarias
- Eucariotas unicelulares
- Células cancerosas
Antes de que una célula se divida, cada cromosoma se duplica (durante la fase S del ciclo celular).
Al inicio de la división celular, los cromosomas duplicados se condensan en estructuras que pueden teñirse con facilidad (por ello denominadas cromosomas), pudiéndose observar bajo el microscopio óptico.
La condensación es tal que el cromosoma es aproximadamente 10.000 veces más corto que la molécula de ADN que contiene .A primera vista, los cromosomas duplicados se mantienen juntos por el centrómero. Mientras están juntos, es común llamar a cada parte del cromosoma duplicado, cromátida hermana.
Esto no debe confundirnos, cada una de las "cromátidas hermanas" es un cromosoma completo. El cinetocoro es una estructura proteica discoidal que forma parte del centrómero y ayuda a separar las cromátidas hermanas. Es el sitio de unión con los microtúbulos del huso, que contienen los motores de dineína que tiran a los cromosomas en la anafase. Además proveen una plataforma para ensamblar y movilizar las proteínas que construyen el huso.
La posición del centrómero, determina el largo de los brazos del cromosoma; en base a esto sepuede clasificar a los cromosomas en:
- Metacéntricos: el centrómero en posición central determina brazos de igual longitud
- Submetacéntricos: un par de brazos es más corto que el otro, pues el centrómero se encuentra alejado del centro.
- Acrocéntricos: el centrómero se halla próximo a uno de los extremos, por lo tanto uno de los brazos es muy pequeño
- Telocéntricos: el centrómero está totalmente desplazado a uno de los extremos, por tanto el otro brazo es prácticamente inexistente

Los cromosomas acrocéntricos poseen una masa de cromatina llamada satélite, en el extremo del brazo corto. El satélite se halla aislado del resto del cromosoma por la constricción secundaria. La zona aledaña al satélite de los cromosomas acrocéntricos contribuye a formar el nucléolo
El más corto de los dos brazos del cromosoma se llama p; el más largo es el brazo q.
Todas las especies tienen un número característico de paresde cromosomas homólogos llamado número diploide (2n). El número diploide del hombre es 46.
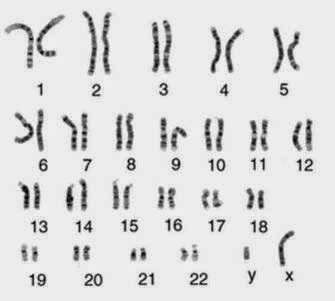



El cariotipo es una representación gráfica o fotográfica de los cromosomas presentes en el núcleo de una sola célula somática de un individuo. Cada miembro del par de cromosomas homólogos proviene de cada uno de los padres del individuo cuyas células examinamos.
El cariotipo de la mujer contiene 23 pares de cromosomas homólogos, 22 pares son autosomas y el par restante, cromosomas sexuales, ambos " X".
El cariotipo del hombre contiene los mismos 22pares de autosomas y 1 par de cromosomas sexuales, un cromosoma sexual "X" y un cromosoma sexual "Y" (un gen en el cromosoma Y designado SRY es el que pone en marcha el desarrollo de un varón, por lo tanto determina el sexo).
El análisis del cariotipo involucra la comparación de cromosomas por su longitud, la ubicación de los centrómeros y la ubicación y los tamaños de las bandas G.
Durante la mitosis, los 23 pares de cromosomas humanos se condensan y son visibles con un microscopio óptico.
Preparación de un Cariotipo: La preparación de un cariotipo normalmente involucra bloquear las células (glóbulos blancos) durante la mitosis con colchicina y marcar los cromosomas condensados con tinción Giemsa. La tinción marca las regiones de los cromosomas que son ricos en pares de nucleótidos entre A -T produciendo una banda oscura, la banda G. Luego de la tinción, los cromosomas se fotografían, se recortan y se ordenan de acuerdo a su longitud. Los de igual tamaño se aparean según la ubicación de su centrómero.




Una error común es suponer que cada banda representa un sólo gen. En realidad las bandas más pequeñas contienen más de un millón de pares de nucleótidos y potencialmente cientos de genes. Por ejemplo, el tamaño de una banda pequeña es igual a toda la información genética de una bacteria.
El análisis del cariotipo es una de muchas técnicas que nos permiten investigar las miles de enfermedades genéticas que sepueden encontrar en los seres humanos.
Cariotipo femenino normal: Los autosomas se ordenan en grupos por tamaño y posición del centrómero.
8. EL NUCLEOLO
En el nucléolo tiene lugar la formación de subunidades ribosómicas, la síntesis y procesamiento de ARNr y actualmente se considera que desempeña un importante papel en la regulación del ciclo celular.
El nucléolo es un aglomerado de fibras de cromatina de distintos cromosomas. En el hombre, los pares 13,14, 15, 21 y 22, aportan sectores de cromatina que forman el nucléolo. Todos estos cromosomas son acrocéntricos y presentan constricciones secundarias denominadas organizadores nucleolares (NOR), donde están los genes que codifican ARNr .




El nucléolo aparececomo una estructura simple carente de componente membranoso, en la que diferenciamos dos regiones:
- Una zona fibrilar central, formada por ADNribosómico y ARNr naciente
- Un zona granular periférica donde los gránulos están formados por las subunidades ribosómicas en proceso de ensamblado (Fig. 10.20).
Los nucléolos, al igual que la envoltura nuclear desaparecen en la mitosis y se reorganizan alrededor de los segmentos de ADNr, que como su nombre lo indica, codifica ARNr. Siendo el ARNr el más abundante dentro de los tipos de ARN, existen múltiples copias del gen que lo codifica. El genoma humano presenta alrededor de 200 copias del gen para ARNr. Estos genes que promedian los 10.000 nucleótidos se localizan en tándem. Cada gen está separado por ADN espaciador y presenta asociado una molécula de ARN polimerasa I. De cada enzima parten perpendicularmente los ARNr nacientes, tomando la apariencia característica de un árbol de navidad. Cada gen produce un transcripto llamado ARNr 45S que será luego procesado.
El tamaño del nucleólo varía entre células y en la misma célula según su atividad, pues si bien la velocidad de transcripción puede acelerarse, el ensamblado de las subunidades ribosomales requiere de un tiempo más o menos constante; es por ello que en los nucléolos grandes observamos mayor proporción de componente granular.
Microfotografía electrónica de los genes nucleolares (ADNr) durante la transcripción. Observe como la longitud de los transcriptos primarios aumenta a medida que nos alejamos del punto de inicio
ANIMACIONES
CONTENIDOS ANIMADOS
9. CICLO VITAL DE LA CELULA.
El ciclo celular (también llamado ciclo de división celular) es una secuencia de sucesos que conducen primeramente alcrecimiento de la célula y posteriormente a la división en células hijas.
El ciclo celular se inicia en el instante en que aparece una nueva célula, descendiente de otra que se ha dividido, y termina en el momento en que dicha célula, por división subsiguiente, origina nuevas células hijas.
Gráfica del ciclo celular y sus etapas o fases.

El ciclo celular es la base para la reproducción de los organismos. Su función no es solamente originar nuevas células sino asegurar que el proceso se realice en forma debida y con la regulación adecuada (con controles internos para evitar la posible creación de células con múltiples errores).
La creación de nuevas células permite al organismo mantenerse en un constante equilibrio, previniendo así aquellos desórdenes que puedan perjudicar su salud (enfermedades congénitas, cáncer, etc.).
Los controles internos en la célula son ejecutados por proteínas que no permiten que se presenten situaciones desastrosas (enfermedades) para un ser vivo.
Las células que no entrarán en división no se consideran que estén en el ciclo celular.
En rigor, el ciclo celular (la secuencia de sucesos) comprende dos periodos bien nítidos: la interfase (etapas G1 – S y G2) y la división celular (etapa M). Esta ultima tiene lugar por mitosis o meiosis.
La interfase es el período comprendido entre divisiones celulares. Es la fase más larga del ciclo celular, ocupando casi el 95 por ciento del ciclo, trascurre entre dos mitosis y como ya vinos se divide en tres subetapas: G1, S y G2.
El estado o etapa G1, del inglés Growth o Gap1 (Intervalo 1), es la primera fase del ciclo celular, en la que existe crecimiento celular con síntesis de proteínas y de ARN. Es el período que trascurre entre el fin de una mitosis y el inicio de la síntesis de ADN. Tiene una duración de entre 6 y 12 horas, y durante este tiempo la célula duplica su tamaño y masa debido a la continua síntesis de todos sus componentes, como resultado de la expresión de los genes que codifican las proteínas responsables de su fenotipoparticular.
El ciclo celular
La división celular, constituida por la mitosis (división del núcleo) y la citocinesis (división del citoplasma), ocurren después de completarse las tres fases preparatorias que constituyen la interfase.
El estado o etapa S (del inglés Synthesis) representa "Síntesis". Es la segunda fase del ciclo, en la que se produce la replicación o síntesis del ADN, como resultado cada cromosoma se duplica y queda formado por dos cromátidas idénticas. Con la duplicación del ADN, el núcleo contiene el doble de proteínas nucleares y de ADN que al principio. Tiene una duración de unos 6-8 horas.
El estado o etapa G2 del inglés Growth o Gap2 (Intervalo 2), es el tiempo que transcurre entre la fase S y el inicio de la mitosis (la célula se prepara para mitosis). Tiene una duración entre 3 y 4 horas. Termina cuando la cromatina empieza a condensarse al inicio de la mitosis.
El estado o etapa M representa “la fase M”, e incluye la mitosis o reparto de material genético nuclear (donde se divide la cromatina duplicada de modo tal que cada célula hija obtenga una copia del material genético o sea un cromosoma de cada tipo) y lacitocinesis (división del citoplasma).
Si el ciclo completo durara 24 horas, la fase M duraría alrededor de media hora (30 minutos).
El final de la mitosis da cabida a un nuevo ciclo en G1 o puede que la célula entre en fase G0 que corresponde a un estado de reposo especial característico de algunas células, en el cual puede permanecer por días, meses y a veces años.
Las células que se encuentran en el ciclo celular se denominanproliferantes y las que se encuentran en fase G0 se llaman células quiescentes.
Aquí es importante recordar que todas las células se originan únicamente de otra existente con anterioridad.
Como todo proceso orgánico, el ciclo celular está sujeto a regulación. Ésta es realizada en sitios específicos llamadospuntos de control o de chequeo, que pueden frenar o disparar diversos procesos que le permitan a la célula proseguir con su ciclo normal de replicación del material genético, crecimiento y división.
La función de la regulación, básicamente es realizada por proteínas específicas conocidas como cinasas y ciclinas (ciclinas A ó B).
Hay células que se encuentran permanentemnete en el ciclo, como las epiteliales; otras están permanentemente fuera del ciclo, como las neuronas, y otras están fuera del ciclo, pero bajo un estímulo adecuado pueden volver adividirse, como es el caso de las células hepáticas.
10. MITOSIS
Durante la fase anterior G1, las moléculas de ADN están enrolladas alrededor de las proteínashistonas, formando los fibras de cromatina que constituyen los cromosomas. Durante la fase S se realiza la duplicación de los cromosomas en el núcleo, por replicación de las moléculas de ADN. Las dos moléculas hijas de cada cromosoma son identicas entre sí, por lo que se denominan cromátidas hermanas.
Durante esta fase se inicia la condensación de los cromatidas hermanas hasta formar loscromosomas mitóticos. Cada cromátida tiene forma de bastocillo y permanecen unidas entre si a través de una región denominada centromero. Según la posición relativa del centrómero en los cromosomas mitóticos, esto se clasifican en:
- Metacéntricos: Centrómero central. Dos brazos iguales en cada cromátida.
- Acrocéntricos: Centrómero distal. Dos brazos desiguales en cada cromátida.
- Telocéntricos: Centrómero terminal. Un solo brazo por cada cromátida.
10.1. FASE M: DIVISION CELULAR
La mitosis es la fase en la cual los nucleos de las células se dividen y se reparten las cromátidas hermanas. Es un proceso continuo en el que se distinguen cinco etapas: Profase, prometafase, metafase, anafase y telofase. Cada etapa viene caracterizada por una serie de cambios celulares.
a) PROFASE: A lo largo de esta fase finaliza la condensación de las cromátidas hermanas iniciada en la fase G2. Los cromosomas mitóticos se van haciendo más cortos y grueos, haciendose visibles al microscopio óptico. En esta fase el complejo centriolar (formado por dos centriolos y fibras pericentriolares), se duplica formando los asteres que comienzan a alejarse a polos opuestos del núcleo, al tiempo que se forman nuevas fibras de microtúbulos polares que constituyen el huso mitótico. Las células animales, que poseen centriolos y por tanto forman aster realizan una mitosis llamda astral, para diferenciarla de la mitosis anastralque realizan las células vegetales que carecen de centriolos. En este caso las fibras del huso se forman en una región del citoplasma cercana al nucleo y libre de orgánulos que se conoce como zona clara.






b) PROMETAFASE: La ruptura de la envoltura nuclear señala el comienzo de esta fase, quedando los cromosomas libres. Se forman los microtúbulos cinetocóricos, a apartir de los cinetocoros de los cromosomas. Estos microtúbulos son perpendiculares al eje del cromosoma. Al final de esta fase los cromosomas comienzan a orientarse respecto a los polos, de modo que cada cinetocoro se encuentra frente a un de los polos.
c) METAFASE. Los cromosomas se disponen con sus cinetocoros en un mismo plano ecuatorial equidistantes de los dos polos, formando la llamada placa metafásica. Los microtúbulos cinetocóricos se alargan haca los polos.






d) ANAFASE. Las cromátidas hermanas de cada cromosoma se separan y se convierten en cromosomas independientes que son desplazdos, a la misma velocidad y simultáneamente, hacia el polo que miran sus cinetocoros. Esto es posible por un acortamiento de los microtúbulos cinetocóricos y un alargamiento de los microtubulos polaraes que alargan el huso y alejan ambos polos.






e) TELOFASE. Al principio desaparecen las microtubulos cinetocóricos y fragmentos de retículo endoplasmático se unen a los cromosomas, formando un esbozo de la envoltura nuclear. Los microtubulos polares se agrupan en haces y se alejan de los polos. Al final de esta fase se completa la envoltura nuclear, los cromosomas comienzan a descondensarse y los microtubulos polares se encuentran agrupados formando un haz único.






10.2. CITOCINESIS.
a) Final de la anafase: la división del citoplasma se inicia al final de la anafase. La membrana citoplasmática se invagina, gracias al anillo contráctil de fibras de actina dispuesto alrededor de la célula, iniciando asi la formación del surco de división.
b) Telofase: Al lo largo de la telofase el surco crece y se comprime alrededor del haz microtúbulos polares
Al final de la telofase las células hijas estan unidas por un puente citoplasmático donde se encuentra el haz de microtúbulos.
c) Separación de las células: El puente citoplasmático se rompe, las celulas se separan y una de ellas arrastra los restos del haz de microtúbulos que finalmente seran dgradados.
En las células vegetales la citocinesis presenta algunas diferencias respecto a lo visto anteriormente:
Durante la telofase, vesículas provenientes del aparato de Golgi confluyen en la región media de la célula y se fusionanan formando el fragmoplasto. Al final de la telofase todas las vesiculas se han fusionado formando las membranas plasmáticas de las dos células hijas. A partir del contenido de la vesiculas se forma la pared celular. Los Puentes o poros persisten entre las dos células y sonstituyen los plasmodesmos.
En general la división citoplasmática puede ser de tres formas diferentes:
- Bipartición: La célula se divide en dos células hijas iguales.
- Gemación: el núcleo se divide una o pocas veces y se separa de la célula madre en forma de yemas.
- División Múltiple: El núcleo se divide muchas veces, cada núcleo se rodea de una porción de citoplasma, quedando libres cuando la membrana de la célula madre se rompe.
El número de cromosomas es igual en todas las células de los individuos de una misma especie, siendo característo de las diferentes especies: Homo sapiens 46 cromosomas, Canis domesticus (perro) 78, Licopersicum sculentum (tomate) 36. Este número de cromosomas se denomina número diploide y se designa 2n, ya que esta formado por pares de cromosomas, denominados cromosomas homólogo. Los pares homologos proceden del gameto masculino y del gameto femenino, y se reunen en el momento de la fecundación. Los gametos poseen por tanto n cromosomas o número haploide. Así, las células del hombre hay 23 parejas de cromososmas homólogos (46), excepto en los gametos que solo poseen 23 cromosomas.
Las células germinativas, a partir de las cuales se originan los gametos, son células diploides. Por tal motivo estas células tienen que sufrir una reducción cromosómica. Esta reducción es posible gracias a la meiosis que consiste basicamente en dos divisiones consecutivas de la célula, con una sola división cromosómica. El resultado es que a partir de una célula 2n se obtienen cuatro células n.
Tal y como ocurre en la mitosis, durante las fases previas S y G1, se produce la duplicación de los cromosomas, los cuales quedan unidos a través del centromero. A partir de aqui la meiosis transcurre en dos divisiones sucesivas:
11.1. Primera División Meiótica.
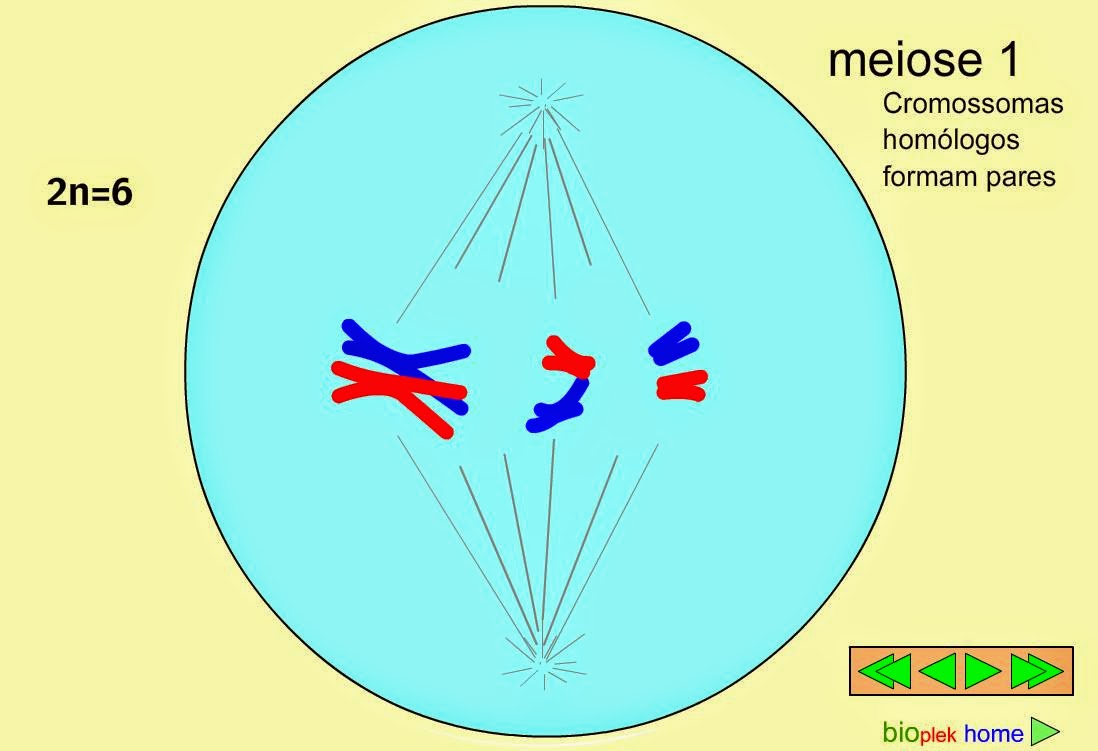
1. PROFASE I.
Es la fase clave de la meiosis y la más larga y compleja. Durante todo el proceso la envoltura nuclear permanece intacta, al tiempo que se desintegran el nucleolo y se forma el huso mitótico. Se distinguen cinco fases:
Es la fase clave de la meiosis y la más larga y compleja. Durante todo el proceso la envoltura nuclear permanece intacta, al tiempo que se desintegran el nucleolo y se forma el huso mitótico. Se distinguen cinco fases:
1.1. Leptoteno: Los cromosomas se hacen visibles por condensación. Sin embargo no se distinguen las cromatidas hermanas pues permanecen estrechamente unidas. Los cromosomas forman bucles de manera que sus extremos se unen a la cara interna de la envoltura nuclear a través de la placa de unión.
1.2. Zigoteno: Los cromosomas homólogos se aparean punto por punto, de forma intima, en toda su longitud. Este emparejamiento gen a gen de los cromosomas homólogos se denominaSinapsis. El apareamiento preciso de los homólogos queda estabilazado y mantenido hasta el final de la siguiente fase gracias a la formación de una estructura proteica en forma de cinta denominada complejo sinaptonémico. La sinapsis se produce entre todos los cromosomas homólogos excepto entre los cromosomas X e Y, que solo se aparean parcialmente.




1.3. Paquiteno: Los cromosomas se encogen haciendose más grueso. Cada par de cromosomas homólogo se denomina bivalente. Durante esta fase se produce el intercambio de fragmentos cromatídico entre cromátidas no hermanas de los cromosomas homólogos, mediante un procesos complejo conocido como sobrecruzamiento y que es la causa de larecombinación genética. Al final del paquiteno son visibles las cromatiadas hermanas, de manera ene cada grupo cromosómico posee cuatro cromátidas y se denomia tetrada.
1.4. Diploteno: Desaparece el complejo sinaptonémico lo que provoca la separación de los cromosomas homólogos. La separación no es total pues las cromátidas no hermanas permanecen unidas por zonas denominadas quiasmas, que son los puntos por donde se produjo el sobrecruzamiento.
1.5. Diacinesis: La condesación de las tetradas es máxima. Las cromátidasno hemanas permanecen unidas por los quiasmas que se han desplazado a los extremos de los cromosomas homólogos. Al final de esta fase comineza la desintegración de la envoltura nuclear y del nucleolo, al tiempo que se duplica el diplosoma, se forma el aster y se inicia la formación del huso con microtúbulos polares
2. METAFASE I.
Se forman los microtúbulos cinetocóricos, pero a diferencia de la mitosis, los dos cinetocoros de un mismo cromosoma homólogo estan orientados hacia el mismo polo, mientras que los cinotocoros del homólogo se orientan hacia el polo opuesto.
3. ANAFASE I.
Los quiasmas se rompesn y al acortarse los microtúbulos cinetocoricos se produce la separación de los cromosomas homólogos, que emigran hacia polos opuestos.
4. TELOFASE I.
Desaparece el huso y se forma la envoltura nuclear. Los cromosomas se sufren una ligera descondensación y la celula se divide en dos.
Como resultado de esta primera división se producen dos células que tiene cada una un juego completo de cromosomas homólogos procedentes de la célula madre. Estos cromosomas homologos ya no son completamente pateno o maternos pues debido al sobrecruzamineto alternan segmentos paternos y maternos al azar. De esta forma la recombinación genética hace que todos los gamentos sean geneticamente diferentes.
11.2. Segunda División Meiótica.
Se trata de un proceso similar a la mitosis. Comprende las fases: profase II, metafase II, anafase II, telofase II y citocinesis que son identicas a las estudiadas en la mitosis . El resultado final de esta segunda división son cuatro células haploides por cada célula madre diploide.
Las consecuencias de la meiosis son por tanto:
a) Reducción a la mitad del número de cromosomas y
b) Obtención de cuatro células diferentes entre sí y diferentes a las progenitoras.
Las células que realizan este proceso se denominan haploides (n cromosomas) y pertenecen a la línea germinal del individuo, frente a las células somáticas, diploides (2n cromosomas), que conservan su doble dotación cromosómica.
DIFERENCIAS ENTRE MITOSIS Y MEIOSIS
| |
MITOSIS
|
MEIOSIS
|
|
|
12. CICLOS BIOLÓGICOS
La meiosis puede llevarse a cabo en diferentes momentos de la vida de los individuos y atendiendo a esta particularidad pueden considerarse tres tipos de organismos y ciclos biológicos: haplontes, diplontes y diplohaplontes.
La meiosis puede llevarse a cabo en diferentes momentos de la vida de los individuos y atendiendo a esta particularidad pueden considerarse tres tipos de organismos y ciclos biológicos: haplontes, diplontes y diplohaplontes.
· En organismos con ciclo haplonte, la meiosis se realiza inmediatamente después de la fecundación y sólo el cigoto es diploide, siendo el individuo adulto haploide. Este tipo de ciclo se presenta en Protoctistas.
En el ciclo diplonte, la meiosis tiene lugar para la formación de los gametos, y estas células son las únicas células haploides y los individuos adultos diploides. Los animales, incluida la especie humana, y ciertas algas son individuos diplontes.

El ciclo diplohaplonte se caracteriza por la alternancia de una fase diploide y otra haploide, por lo que se llama también reproducción alternante. La meiosis y la fecundación se realizan en momentos muy separados. Todos los vegetales presentan este tipo de ciclo biológico.

Se piensa que el ciclo más primitivo es el haplonte debido a que lo presentan los organismos menos evolucionados. Precisamente a lo largo de la evolución se ha ido aplazando el momento en el que se realiza la meiosis, lo cual representa una ventaja evolutiva y es que todas las células del individuo, excepto los gametos, son diploides por lo que poseen una doble información genética para cada carácter; si se deteriora una de estas informaciones, todavía queda la otra; esto hace que se incremente la estabilidad genética de los individuos y explica la amplia distribución de las especies diploides.
ANIMACIONES CICLO CELULAR
ANIMACIONES MITOSIS
ANIMACIONES MEIOSIS
Cada vez que una célula se reproduce por división su material genético debe ser copiado para que las dos células hijas dispongan una copia completa del mismo. Uno de los requisitos que debería cumplir una eventual molécula de la herencia, es precisamente la capacidad para crear copias fieles de sí misma de manera que la información pudiese ser transmitida en las sucesivas generaciones celulares.
Watson y Crick se percataron, y así lo sugerían en su artículo de Nature, de que el modelo de estructura en doble hélice que habían elaborado proporcionaba las bases físico-químicas para un mecanismo de replicación del DNA.
En efecto, la estricta complementariedad de bases nitrogenadas a lo largo de la doble hélice permite que cada una de las dos cadenas polinucleotídicas que la forman pueda ser utilizada como molde para sintetizar una nueva cadena complementaria. Así, desenrollando las dos cadenas de una hélice original y utilizando cada una de ellas como molde para sintetizar una nueva cadena, en la que los nucleótidos se irán añadiendo conforme a las reglas que rigen el emparejamiento de las bases, se obtendrán dos dobles hélices “hijas” idénticas, portadoras de la misma información. El mecanismo que aquí se ha esbozado fue propuesto por Watson y Crick en un artículo publicado en el número de Nature de mayo de 1953, aclarando así lo que sugerían en su artículo del mes anterior. Tal mecanismo fue conocido como replicación semiconservativa para resaltar el hecho de que cada una de las dobles hélices hijas conserva la mitad, es decir, una de las cadenas, de la doble hélice original.
El modelo de replicación semiconservativa propuesta por Watson y Crick pareció acertado a muchos investigadores por su sencillez y elegancia. Sin embargo, se plantearon algunos modelos alternativos que no podían ser descartados a priori. Uno de ellos era el modelo de replicación conservativa, según el cual la doble hélice original, sin perder su integridad, sirve de patrón para sintetizar una doble hélice hija totalmente nueva, de manera que la doble hélice original se conserva intacta a lo largo de las sucesivas generaciones celulares. Otro era el modelo de replicación dispersiva, según el cual la doble hélice original se descompone en infinidad de pequeños fragmentos, cada uno de los cuales sirve de molde para sintetizar un fragmento complementario siguiendo las reglas de emparejamiento de bases; a continuación los fragmentos resultantes se empalman en el orden correcto para dar lugar a dos dobles hélices hijas cada una de las cuales contiene fragmentos de la original y fragmentos de nueva síntesis.
14. EL EXPERIMENTO DE MESELSON Y STAHL
Un brillante experimento realizado por M. S. Meselson y F. W. Stahl en 1957 demostró que el modelo semiconservativo propuesto por Watson Y Crick era el correcto. El diseño experimental se basó en el uso de dos isótopos estables del nitrógeno: el isótopo más abundante en la naturaleza (14N) y un isótopo pesado (15N), más escaso.
El modelo de replicación semiconservativa propuesta por Watson y Crick pareció acertado a muchos investigadores por su sencillez y elegancia. Sin embargo, se plantearon algunos modelos alternativos que no podían ser descartados a priori. Uno de ellos era el modelo de replicación conservativa, según el cual la doble hélice original, sin perder su integridad, sirve de patrón para sintetizar una doble hélice hija totalmente nueva, de manera que la doble hélice original se conserva intacta a lo largo de las sucesivas generaciones celulares. Otro era el modelo de replicación dispersiva, según el cual la doble hélice original se descompone en infinidad de pequeños fragmentos, cada uno de los cuales sirve de molde para sintetizar un fragmento complementario siguiendo las reglas de emparejamiento de bases; a continuación los fragmentos resultantes se empalman en el orden correcto para dar lugar a dos dobles hélices hijas cada una de las cuales contiene fragmentos de la original y fragmentos de nueva síntesis.
Un brillante experimento realizado por M. S. Meselson y F. W. Stahl en 1957 demostró que el modelo semiconservativo propuesto por Watson Y Crick era el correcto. El diseño experimental se basó en el uso de dos isótopos estables del nitrógeno: el isótopo más abundante en la naturaleza (14N) y un isótopo pesado (15N), más escaso.
En primer lugar Meselson y Stahl dispusieron dos cultivos de la bacteria E. coli creciendo sobre un medio en el que la única fuente de nitrógeno era NH4Cl. En uno de los cultivos el NH4Cl contenía el isótopo normal del nitrógeno (14N) y en el otro el isótopo pesado (15N). De este modo, el isótopo correspondiente a cada medio de cultivo se incorporaba a todas las biomoléculas nitrogenadas de las bacterias que crecían en él, incluyendo los ácidos nucleicos. A continuación extrajeron el DNA de las células de uno y otro cultivo, lo mezclaron y lo sometieron a centrifugación a alta velocidad en un gradiente de densidad de CsCl. Así comprobaron que el DNA “ligero” y el DNA “pesado” que habían incorporado uno u otro isótopo se situaban en dos bandas perfectamente distinguibles en el tubo de la centrífuga.
En una segunda fase de su experimento Meselson y Stahl transfirieron bacterias que habían estado reproduciéndose durante varias generaciones en un medio de cultivo con nitrógeno pesado a un medio de cultivo con nitrógeno ligero, de manera que, a partir del instante de la transferencia, todo el DNA que se sintetizase en las células transferidas incorporaría exclusivamente nitrógeno ligero. Seguidamente, a intervalos regulares de 20 minutos (equivalentes al período de generación de las bacterias) extrajeron el DNA de sucesivas muestras del cultivo y lo sometieron a centrifugación en gradiente de densidad de CsCl.

En efecto, si el modelo de replicación fuese semiconservativo como habían predicho Watson y Crick, todo el DNA extraído tras los 20 primeros minutos consistiría en dobles hélices formadas por una cadena ligera (recién sintetizada) y otra cadena pesada (la original), lo que se traduciría en un DNA de densidad “híbrida” que en el tubo de la centrífuga ocuparía una posición intermedia entre las correspondientes al DNA “pesado” y el “ligero”.
En una segunda fase de su experimento Meselson y Stahl transfirieron bacterias que habían estado reproduciéndose durante varias generaciones en un medio de cultivo con nitrógeno pesado a un medio de cultivo con nitrógeno ligero, de manera que, a partir del instante de la transferencia, todo el DNA que se sintetizase en las células transferidas incorporaría exclusivamente nitrógeno ligero. Seguidamente, a intervalos regulares de 20 minutos (equivalentes al período de generación de las bacterias) extrajeron el DNA de sucesivas muestras del cultivo y lo sometieron a centrifugación en gradiente de densidad de CsCl.

El éxito del experimento de Meselson y Stahl radicó en que su diseño permitía formular predicciones sobre los resultados en función de cada uno de los tres modelos de replicación propuestos, siendo los resultados predichos perfectamente distinguibles y excluyentes entre sí.


Transcurridos otros 20 minutos, es decir, en la segunda generación de bacterias tras la transferencia, la mitad de las dobles hélices serían de densidad híbrida mientras que la otra mitad serían totalmente ligeras, dando lugar en el tubo de la centrífuga a dos bandas en las posiciones correspondientes.
Si por el contrario la replicación respondiese a un modelo conservativo nunca aparecerían bandas híbridas, pues el DNA de las bacterias de la primera generación estaría compuesto por dobles hélices la mitad de las cuales serían totalmente ligeras (las recién sintetizadas) y la otra mitad totalmente pesadas (las originales), de manera que en el tubo de la centrífuga aparecería una banda ligera y otra pesada. Lo mismo ocurriría en la segunda generación de bacterias, con la única diferencia de que la proporción de hélices ligeras respecto a hélices pesadas sería de 3:1 en lugar de 1:1, lo que se traduciría en una mayor densidad de moléculas de DNA ligero en la banda correspondiente.
En el caso de que la replicación siguiese un modelo dispersivo los resultados obtenidos en la primera generación serían iguales a los que predice el modelo semiconservativo: el DNA extraído de las bacterias de la primera generación estaría formado por fragmentos ligeros y fragmentos pesados que se repartirían en una y otra cadena polinucleotídica de la doble hélice, y ello daría lugar a la aparición de una única banda en el tubo de la centrífuga que ocuparía la posición correspondiente al DNA de densidad híbrida. Esta banda híbrida persistiría en la segunda y sucesivas generaciones, aunque cabría esperar que tras varios ciclos de replicación, al ir aumentando la proporción de fragmentos ligeros con respecto a los pesados, empezasen a aparecer dobles hélices totalmente ligeras que irían formando una banda en su posición correspondiente.
Los resultados obtenidos por Meselson y Stahl se ajustaban con precisión a los predichos por el modelo de replicación semiconservativa. No obstante, dado que también eran parcialmente compatibles con el modelo dispersivo (al menos en lo que se refiere a las bandas obtenidas con el DNA de la primera generación), realizaron una posterior comprobación con el objeto de descartar definitivamente este modelo. Tomaron una nueva muestra de DNA de las bacterias de la primera generación y, antes de centrifugarlo, procedieron a desnaturalizarlo. Así, al separar físicamente las dos cadenas polinucleotídicas que integran la doble hélice deberían obtenerse dos bandas (una ligera y otra pesada) en el tubo de la centrífuga en el caso de que la replicación fuese semiconservativa, o bien una sola banda híbrida en el caso de que fuese dispersiva. Los resultados de este último experimento no dejaban lugar a dudas: Watson y Crick habían acertado también con el mecanismo que rige la replicación del DNA.
ANIMACIONES

15. OTRAS CARACTERÍSTICAS DE LA REPLICACION
Una vez demostrado el carácter semiconservativo de la replicación del DNA se plantearon nuevos interrogantes acerca de algunos aspectos del proceso. En primer lugar cabe preguntarse si en la replicación de una gran molécula de DNA ¿se desenrollan y separan primero las dos cadenas polinucleotídicas en toda su longitud para luego servir cada una de ellas como molde para la síntesis de su complementaria?, o bien ¿los procesos de separación de las cadenas y la síntesis de sus complementarias están de alguna manera acopladas en el espacio y en el tiempo?
Los estudios realizados demostraron que la replicación del DNA es ordenada y secuencial, es decir, se inicia en unos puntos fijos del cromosoma, y el crecimiento de las nuevas cadenas de DNA se produce de manera simultánea al desenrollamiento de la doble hélice original.
Para elucidar este y otros aspectos de la replicación del DNA se utilizó una técnica basada en isótopos radiactivos denominada autorradiografía. En un cultivo de E. coli introdujo en el medio de cultivo timina tritiada (timina en la que los átomos de hidrógeno habían sido sustituidos por su isótopo radiactivo 3H), de manera que esta base nitrogenada se incorporaba a las cadenas de DNA en crecimiento.
Si por el contrario la replicación respondiese a un modelo conservativo nunca aparecerían bandas híbridas, pues el DNA de las bacterias de la primera generación estaría compuesto por dobles hélices la mitad de las cuales serían totalmente ligeras (las recién sintetizadas) y la otra mitad totalmente pesadas (las originales), de manera que en el tubo de la centrífuga aparecería una banda ligera y otra pesada. Lo mismo ocurriría en la segunda generación de bacterias, con la única diferencia de que la proporción de hélices ligeras respecto a hélices pesadas sería de 3:1 en lugar de 1:1, lo que se traduciría en una mayor densidad de moléculas de DNA ligero en la banda correspondiente.
En el caso de que la replicación siguiese un modelo dispersivo los resultados obtenidos en la primera generación serían iguales a los que predice el modelo semiconservativo: el DNA extraído de las bacterias de la primera generación estaría formado por fragmentos ligeros y fragmentos pesados que se repartirían en una y otra cadena polinucleotídica de la doble hélice, y ello daría lugar a la aparición de una única banda en el tubo de la centrífuga que ocuparía la posición correspondiente al DNA de densidad híbrida. Esta banda híbrida persistiría en la segunda y sucesivas generaciones, aunque cabría esperar que tras varios ciclos de replicación, al ir aumentando la proporción de fragmentos ligeros con respecto a los pesados, empezasen a aparecer dobles hélices totalmente ligeras que irían formando una banda en su posición correspondiente.
Los resultados obtenidos por Meselson y Stahl se ajustaban con precisión a los predichos por el modelo de replicación semiconservativa. No obstante, dado que también eran parcialmente compatibles con el modelo dispersivo (al menos en lo que se refiere a las bandas obtenidas con el DNA de la primera generación), realizaron una posterior comprobación con el objeto de descartar definitivamente este modelo. Tomaron una nueva muestra de DNA de las bacterias de la primera generación y, antes de centrifugarlo, procedieron a desnaturalizarlo. Así, al separar físicamente las dos cadenas polinucleotídicas que integran la doble hélice deberían obtenerse dos bandas (una ligera y otra pesada) en el tubo de la centrífuga en el caso de que la replicación fuese semiconservativa, o bien una sola banda híbrida en el caso de que fuese dispersiva. Los resultados de este último experimento no dejaban lugar a dudas: Watson y Crick habían acertado también con el mecanismo que rige la replicación del DNA.
ANIMACIONES

15. OTRAS CARACTERÍSTICAS DE LA REPLICACION
Una vez demostrado el carácter semiconservativo de la replicación del DNA se plantearon nuevos interrogantes acerca de algunos aspectos del proceso. En primer lugar cabe preguntarse si en la replicación de una gran molécula de DNA ¿se desenrollan y separan primero las dos cadenas polinucleotídicas en toda su longitud para luego servir cada una de ellas como molde para la síntesis de su complementaria?, o bien ¿los procesos de separación de las cadenas y la síntesis de sus complementarias están de alguna manera acopladas en el espacio y en el tiempo?
Los estudios realizados demostraron que la replicación del DNA es ordenada y secuencial, es decir, se inicia en unos puntos fijos del cromosoma, y el crecimiento de las nuevas cadenas de DNA se produce de manera simultánea al desenrollamiento de la doble hélice original.
Para elucidar este y otros aspectos de la replicación del DNA se utilizó una técnica basada en isótopos radiactivos denominada autorradiografía. En un cultivo de E. coli introdujo en el medio de cultivo timina tritiada (timina en la que los átomos de hidrógeno habían sido sustituidos por su isótopo radiactivo 3H), de manera que esta base nitrogenada se incorporaba a las cadenas de DNA en crecimiento.
Transcurridas dos generaciones procedió a la extracción del DNA bacteriano mediante una técnica que respetaba la integridad de las moléculas extraídas (lisis de la pared celular con enzimas específicos) y permitió que estas moléculas sedimentasen lentamente sobre un papel de filtro que después fue cuidadosamente montado sobre una placa con emulsión fotosensible.
La placa se guardó en la oscuridad durante dos meses y a continuación se procedió a su revelado. Las emisiones β de los átomos de tritio incorporados al DNA en los restos de timina habían impresionado la placa fotográfica creando unas figuras que se correspondían con cromosomas bacterianos en distintos estadios de replicación. El análisis de estas imágenes, además de proporcionar la primera prueba directa del carácter circular del cromosoma bacteriano, condujo a la conclusión de que la replicación comienza en un único origen, en el que se forma una única “burbuja de replicación” que va creciendo a medida que el proceso avanza.


Posteriores refinamientos de la técnica de autorradiografía, consistentes en el uso de “pulsos” de timina tritiada de pocos segundos de duración, permitieron obtener imágenes de las zonas del cromosoma en las que está teniendo lugar la replicación del DNA en un instante dado. El análisis de estas imágenes reveló que el proceso de replicación es bidireccional: dos horquillas de replicación avanzan simultáneamente en direcciones opuestas partiendo de un único origen.
Años más tarde, usando las mismas técnicas, se pudo comprobar que en los cromosomas eucariotas la replicación se atiene a los mismos principios que en los procariotas, con la excepción de que en aquéllos existen varios orígenes de replicación que dan lugar a varios ojos de replicación por cromosoma. Estos ojos de replicación crecen de manera bidireccional hasta que se funden con sus vecinos completando el proceso.


Años más tarde, usando las mismas técnicas, se pudo comprobar que en los cromosomas eucariotas la replicación se atiene a los mismos principios que en los procariotas, con la excepción de que en aquéllos existen varios orígenes de replicación que dan lugar a varios ojos de replicación por cromosoma. Estos ojos de replicación crecen de manera bidireccional hasta que se funden con sus vecinos completando el proceso.


16. REPLICACIÓN DEL ADN
El proceso de replicación del DNA implica la adición de nucleótidos a una cadena polinucleotídica en crecimiento complementaria de la que le sirve como molde. Los sucesivos nucleótidos deben establecer enlaces fosfodiéster con los restos precedentes y la formación de estos enlaces requiere energía. Debido a ello, los nuevos nucleótidos que se incorporan al proceso no pueden ser nucleótidos monofosfato sino que las células deben recurrir a nucleótidos trifosfato, mucho más energéticos, cuya escisión pirofosforolítica proporciona la energía necesaria para la formación de dichos enlaces. La incorporación de un resto nucleotídico a la cadena en crecimiento supone pues la siguiente reacción química:
ADN (n) + dNTP à ADN (n+1) + PPi
Es conveniente resaltar que esta reacción química, como la mayoría de las que ocurren en las células vivas, es reversible, por lo que altas concentraciones de pirofosfato inorgánico podrían hacer revertir esta reacción hacia la degradación (pirofosforólisis) del DNA celular, con la consiguiente muerte de la célula. Por ello, las células vivas disponen de un enzima de gran eficacia y ubicuidad, llamado pirofosfatasa inorgánica, que degrada rápidamente el pirofosfato (PPi) a ortofosfato (2 Pi) con el objeto de preservar la integridad de los ácidos nucleicos celulares a pesar del considerable gasto energético que esta degradación supone.
La reacción de polimerización del ADN, como todas las reacciones químicas celulares requiere de la actuación de enzimas específicos que catalicen y controlen este proceso. A mediados de los años 50 se inicaron las investigaciones acerca de la síntesis enzimática del DNA en la bacteria E. coli. Pronto se consiguió identificar y aislar un enzima capaz de catalizar la reacción de polimerización que fue denominado ADN polimerasa I.
Estudios posteriores pusieron de manifiesto que el proceso de replicación presentaba una gran complejidad y que era necesaria la concurrencia de toda una batería de enzimas y otras proteínas no enzimátitcas para que culminase con éxito.
El equipo de enzimas y proteínas no enzimáticas implicados en la síntesis del DNA son:
Proteínas ADN A. Relajan la doble hélice en la zona donde ha de iniciarse la replicación preparándola para la acción de las siguientes proteínas.
Helicasas. Su actividad consiste en desenrollar las dos cadenas de la doble hélice haciéndolas girar alrededor del eje central. En el inicio del proceso dan lugar a la llamada “burbuja” u “ojal” de replicación. A partir del origen dos equipos de helicasas se desplazan en sentidos opuestos haciendo avanzar las dos horquillas de replicación.
Proteínas SSB. Moléculas que se unen en gran número a las cadenas sencillas de DNA y las estabilizan con el objeto de evitar que vuelvan a asociarse y que se cierre así la burbuja de replicación.
Girasas y topoisomerasas. El desenrrollamiento de la doble hélice por acción de las helicasas genera tensiones en la molécula de DNA que tiende a formar “superenrrollamientos” del tipo de los que aparecen en el cable de un auricular telefónico cuando inadvertidamente lo hacemos girar sobre sí mismo. Para relajar estas tensiones unos enzimas llamados topoisomerasas cortan de trecho en trecho algún enlace fosfodiéster de la cadena molde y lo vuelven a soldar después de que otras proteínas, las girasas, liberan la tensión mediante el giro del extremo que queda temporalmente libre.
Exonucleasas. Enzimas que escinden nucleótidos a partir del extremo de una cadena polinucleotídica. Unas actúan en dirección 5’ à 3’ y otras lo hacen en dirección 3’ à 5’. Su función es eliminar fragmentos de cadenas polinucleotídicas en algunas fases del proceso de replicación.
ADN Polimerasas. Enzimas que añaden nucleótidos a una cadena de DNA en crecimiento que es complementaria de otra que utilizan como molde. Existen varios tipos de DNA polimerasas que difieren en sus características y actividades. En células procariotas se han identificado 3 ADN polimerasas (I, II y III) mientras que en células eucariotas se han identificado varias (α, β, γ, δ, ε). Todas las ADN polimerasas conocidas añaden nucleótidos a una cadena que crece en dirección 5’ à 3` recorriendo el molde en dirección 3’ à 5’, es decir, catalizan la condensación del grupo 3’ OH libre del último nucleótido de la cadena con el grupo α-5’fosfato del nucleótido que se añade.
El equipo de enzimas y proteínas no enzimáticas implicados en la síntesis del DNA son:
Proteínas ADN A. Relajan la doble hélice en la zona donde ha de iniciarse la replicación preparándola para la acción de las siguientes proteínas.
Helicasas. Su actividad consiste en desenrollar las dos cadenas de la doble hélice haciéndolas girar alrededor del eje central. En el inicio del proceso dan lugar a la llamada “burbuja” u “ojal” de replicación. A partir del origen dos equipos de helicasas se desplazan en sentidos opuestos haciendo avanzar las dos horquillas de replicación.
Proteínas SSB. Moléculas que se unen en gran número a las cadenas sencillas de DNA y las estabilizan con el objeto de evitar que vuelvan a asociarse y que se cierre así la burbuja de replicación.
Girasas y topoisomerasas. El desenrrollamiento de la doble hélice por acción de las helicasas genera tensiones en la molécula de DNA que tiende a formar “superenrrollamientos” del tipo de los que aparecen en el cable de un auricular telefónico cuando inadvertidamente lo hacemos girar sobre sí mismo. Para relajar estas tensiones unos enzimas llamados topoisomerasas cortan de trecho en trecho algún enlace fosfodiéster de la cadena molde y lo vuelven a soldar después de que otras proteínas, las girasas, liberan la tensión mediante el giro del extremo que queda temporalmente libre.
Exonucleasas. Enzimas que escinden nucleótidos a partir del extremo de una cadena polinucleotídica. Unas actúan en dirección 5’ à 3’ y otras lo hacen en dirección 3’ à 5’. Su función es eliminar fragmentos de cadenas polinucleotídicas en algunas fases del proceso de replicación.
ADN Polimerasas. Enzimas que añaden nucleótidos a una cadena de DNA en crecimiento que es complementaria de otra que utilizan como molde. Existen varios tipos de DNA polimerasas que difieren en sus características y actividades. En células procariotas se han identificado 3 ADN polimerasas (I, II y III) mientras que en células eucariotas se han identificado varias (α, β, γ, δ, ε). Todas las ADN polimerasas conocidas añaden nucleótidos a una cadena que crece en dirección 5’ à 3` recorriendo el molde en dirección 3’ à 5’, es decir, catalizan la condensación del grupo 3’ OH libre del último nucleótido de la cadena con el grupo α-5’fosfato del nucleótido que se añade.



Por otra parte, todas necesitan de una cadena que actúe como molde, es decir, no pueden añadir nucleótidos sin más a una cadena polinucleotídica simple. Otro rasgo importante de todas las ADN polimerasas es que todas ellas pueden prolongar una cadena polinucleotídica pero ninguna de ellas puede iniciarlas, es decir, no pueden colocar el primer nucleótido de la cadena sino que precisan de un tramo preexistente, que en lo sucesivo llamaremos primer o cebador, al que añadir nuevos nucleótidos.
Primasas. Enzimas capaces de sintetizar una cadena corta de ARN utilizando un molde de ADN (en realidad son ARN polimerasas-ADN dirigidas). A diferencia de las DNA polimerasas, las primasas sí pueden iniciar cadenas. Son las encargadas de crear los cebadores de ARN sobre los que después han de actuar las ADN polimerasas.
ADN Ligasas. Enzimas que establecen puentes fosfodiéster entre nucleótidos adyacentes que se encuentran en fragmentos diferentes de cadena polinucleotídica. Son necesarias en distintos momentos del proceso replicativo para “soldar” los fragmentos sueltos.
Además de los descritos existen algunos enzimas mixtos, que en una sola molécula presentan más de una actividad. Por ejemplo, las DNA polimerasas presentan en general actividad de exonucleasa en una o en ambas direcciones de la cadena.
Por ser el más estudiado, adoptaremos como modelo el proceso de replicación en la bacteria E. coli, que con muy pequeñas modificaciones es extensible a todas las células procariotas. Seguidamente se analizarán algunas particularidades que presenta el proceso replicativo en las células eucariotas. Distinguiremos en el proceso tres fases: iniciación, elongación y terminación.
16.1. Fase de iniciaciónLa replicación del DNA se inicia en un lugar concreto del cromosoma denominado origen de replicación. Existe un único origen por cromosoma en todas las especies bacterianas conocidas. El origen de replicación consiste en un tramo de 245 pares de base de longitud, conocido como oriC,que contiene varias repeticiones en serie de secuencias específicas de nucleótidos que han podido ser identificadas. Estas secuencias son ricas en pares A-T, lo que facilita la apertura de la doble hélice, y son reconocibles por proteínas específicas que se fijan a ellas.
Un ciclo de replicación comienza cuando varias unidades monoméricas de la proteína específica ADN A se fijan sobre las correspondientes secuencias específicas repetidas de 9 pares de bases de longitud dentro del tramo oriC. A continuación se van añadiendo nuevas unidades monoméricas de ADN A que forman un complejo nucleoproteico con unas veinte de estas unidades y el tramo oriC de la doble hélice de ADN que se dispone formando un bucle alrededor de ellas.
Primasas. Enzimas capaces de sintetizar una cadena corta de ARN utilizando un molde de ADN (en realidad son ARN polimerasas-ADN dirigidas). A diferencia de las DNA polimerasas, las primasas sí pueden iniciar cadenas. Son las encargadas de crear los cebadores de ARN sobre los que después han de actuar las ADN polimerasas.
ADN Ligasas. Enzimas que establecen puentes fosfodiéster entre nucleótidos adyacentes que se encuentran en fragmentos diferentes de cadena polinucleotídica. Son necesarias en distintos momentos del proceso replicativo para “soldar” los fragmentos sueltos.
Además de los descritos existen algunos enzimas mixtos, que en una sola molécula presentan más de una actividad. Por ejemplo, las DNA polimerasas presentan en general actividad de exonucleasa en una o en ambas direcciones de la cadena.
Por ser el más estudiado, adoptaremos como modelo el proceso de replicación en la bacteria E. coli, que con muy pequeñas modificaciones es extensible a todas las células procariotas. Seguidamente se analizarán algunas particularidades que presenta el proceso replicativo en las células eucariotas. Distinguiremos en el proceso tres fases: iniciación, elongación y terminación.
16.1. Fase de iniciaciónLa replicación del DNA se inicia en un lugar concreto del cromosoma denominado origen de replicación. Existe un único origen por cromosoma en todas las especies bacterianas conocidas. El origen de replicación consiste en un tramo de 245 pares de base de longitud, conocido como oriC,que contiene varias repeticiones en serie de secuencias específicas de nucleótidos que han podido ser identificadas. Estas secuencias son ricas en pares A-T, lo que facilita la apertura de la doble hélice, y son reconocibles por proteínas específicas que se fijan a ellas.
Un ciclo de replicación comienza cuando varias unidades monoméricas de la proteína específica ADN A se fijan sobre las correspondientes secuencias específicas repetidas de 9 pares de bases de longitud dentro del tramo oriC. A continuación se van añadiendo nuevas unidades monoméricas de ADN A que forman un complejo nucleoproteico con unas veinte de estas unidades y el tramo oriC de la doble hélice de ADN que se dispone formando un bucle alrededor de ellas.
Algunos de los monómeros de ADN A quedan así situados sobre las secuencias específicas repetitivas de 13 pares de bases de longitud que se encuentran en el otro extremo de oriC, provocando en ellas una desestabilización de la doble hélice y la consiguiente apertura de la burbuja de replicación. La proteína ADN A tiene la función de reconocer el origen de replicación, provocar la apertura de la burbuja y facilitar la entrada de las demás proteínas que forman parte del complejo de iniciación.
Una vez abierta la burbuja de replicación se introducen en ella las proteínas denominadas DNA B y ADN C. La primera de ellas tiene actividad helicasa y actúa agrandando la burbuja de replicación (dos moléculas de ADN B actúan en direcciones opuestas haciendo avanzar las recién creadas horquillas). La presencia de ADN C se requiere únicamente para que ADN B inicie su actuación, abandonando el complejo inmediatamente después. Por el contrario, la helicasa ADN B, permanece en su lugar y sigue actuando también durante la fase de elongación.

Una vez abierta la burbuja de replicación se introducen en ella las proteínas denominadas DNA B y ADN C. La primera de ellas tiene actividad helicasa y actúa agrandando la burbuja de replicación (dos moléculas de ADN B actúan en direcciones opuestas haciendo avanzar las recién creadas horquillas). La presencia de ADN C se requiere únicamente para que ADN B inicie su actuación, abandonando el complejo inmediatamente después. Por el contrario, la helicasa ADN B, permanece en su lugar y sigue actuando también durante la fase de elongación.

A medida que la burbuja de replicación se va haciendo mayor van quedando expuestos tramos cada vez mayores de ADN de cadena sencilla. A estos tramos se unen las proteínas SSB con el objeto de protegerlos del ataque de nucleasas que podrían degradarlos y de evitar que se reconstruya la doble hélice cerrándose con ello la propia burbuja de replicación.
Dado que no existe ninguna ADN polimerasa capaz de iniciar cadenas polinucleotídicas se hace necesaria ahora la concurrencia de una primasa (proteína ADN G) que inicia una cadena corta de RNA (unos 12 nucleótidos de longitud) que actuará como cebador al que la ADN polimerasa podrá añadir nucleótidos en la fase de elongación. La primasa ADN G junto con la helicasa ADN B forman una unidad funcional denominada primosoma cuya integridad es necesaria para que la iniciación tenga lugar.
16.2. Fase de elongación
La fase de elongación comienza con la progresión en ambas direcciones de las horquillas de replicación mediante la acción de las helicasas, que van desenrollando la doble hélice. La acción combinada de girasa y topoisomerasa en ambas horquillas va relajando las tensiones asociadas al superenrollamiento. Mientras tanto, las proteínas SSB van estabilizando los tramos de cadena sencilla que quedan temporalmente expuestos. Todas estas actuaciones se prolongan durante toda la fase de elongación.
Es ahora cuando se pone de manifiesto un problema técnico que afecta a todo el proceso de replicación y que las células vivas, en el transcurso de su evolución, se han visto obligadas a solucionar. Dado que las dos cadenas polinucleotídicas de una doble hélice son antiparalelas, cuando una horquilla de replicación avanza, lo hace en la dirección 5’ à 3’ para una de las cadenas y en dirección 3’ à 5’ para la otra. Por otra parte, se ha comentado ya que todas las Adn polimerasas conocidas añaden nucleótidos a una cadena que crece en dirección 5’ à 3’ mientras que recorren el molde en la dirección opuesta.
Dado que no existe ninguna ADN polimerasa capaz de iniciar cadenas polinucleotídicas se hace necesaria ahora la concurrencia de una primasa (proteína ADN G) que inicia una cadena corta de RNA (unos 12 nucleótidos de longitud) que actuará como cebador al que la ADN polimerasa podrá añadir nucleótidos en la fase de elongación. La primasa ADN G junto con la helicasa ADN B forman una unidad funcional denominada primosoma cuya integridad es necesaria para que la iniciación tenga lugar.
16.2. Fase de elongación
La fase de elongación comienza con la progresión en ambas direcciones de las horquillas de replicación mediante la acción de las helicasas, que van desenrollando la doble hélice. La acción combinada de girasa y topoisomerasa en ambas horquillas va relajando las tensiones asociadas al superenrollamiento. Mientras tanto, las proteínas SSB van estabilizando los tramos de cadena sencilla que quedan temporalmente expuestos. Todas estas actuaciones se prolongan durante toda la fase de elongación.
Es ahora cuando se pone de manifiesto un problema técnico que afecta a todo el proceso de replicación y que las células vivas, en el transcurso de su evolución, se han visto obligadas a solucionar. Dado que las dos cadenas polinucleotídicas de una doble hélice son antiparalelas, cuando una horquilla de replicación avanza, lo hace en la dirección 5’ à 3’ para una de las cadenas y en dirección 3’ à 5’ para la otra. Por otra parte, se ha comentado ya que todas las Adn polimerasas conocidas añaden nucleótidos a una cadena que crece en dirección 5’ à 3’ mientras que recorren el molde en la dirección opuesta.
Por todo ello, la síntesis de DNA sólo puede acompañar al avance de una horquilla de replicación en una de las cadenas (la que la DNA polimerasa recorre como molde en dirección 3’ à 5’). ¿Qué ocurre entonces con la otra cadena? El investigador japonés R. Okazaki demostró en 1968, utilizando el marcaje radiactivo con timina tritiada, que la síntesis en esta otra cadena es discontinua: las DNA polimerasas incorporan nuevos nucleótidos en dirección contraria a la de avance de la horquilla generando unos fragmentos de ADN de 1.000 a 2.000 nucleótidos de longitud, denominados fragmentos de Okazaki.
La cadena en la que la síntesis es continua se denomina hebra conductora y aquella en la que la síntesis es discontinua se denomina hebra rezagada. Como resulta evidente, el inicio de cada fragmento debe aguardar a que la horquilla avance la longitud correspondiente para luego ir creciendo en dirección contraria a dicho avance. Okazaki y sus colaboradores demostraron en 1972 que cada fragmento recién formado lleva unido su correspondiente cebador que deberá ser después eliminado.
DNA pol I
|
DNA pol II
|
DNA pol III
| |
Actividad polimerasa
|
SI
|
SI
|
SI
|
Actividad
Exonucleasa 3’→5’
|
SI
|
SI
|
NO
|
Actividad
Exonucleasa 5’→3’
|
SI
|
NO
|
NO
|
Función biológica
|
Replicación DNA
Eliminación cebadores
|
Reparación DNA
|
Replicación DNA
|
| Tabla 19.1 | |||
Una vez colocados los dos primeros cebadores por la primasa, la DNA polimerasa III comienza a añadirles nucleótidos en dirección 5’à3’ iniciando así las hebras conductoras de ambas horquillas de replicación. Nos referiremos en lo sucesivo a lo que ocurre en una de estas horquillas, considerando que los procesos que ocurren en la otra son análogos. Cuando la hebra conductora ha avanzado entre 1.000 y 2.000 nucleótidos la primasa coloca el cebador del primer fragmento de Okazaki, que a continuación es prolongado por la DNA polimerasa III hasta que se encuentra con el cebador de la hebra conductora de la otra horquilla de replicación y ahí se desprende.


Este proceso se repite cuantas veces sea necesario, a medida que el equipo de helicasas, girasa y topoisomerasava abriendo la doble hélice, hasta completar la replicación del cromosoma. Un fragmento de Okazaki es colocado en la hebra rezagada en cada ciclo mientras que la hebra conductora se sintetiza sin interrupción; cada fragmento finaliza cuando la DNA polimerasa III se encuentra con el cebador del fragmento precedente, momento en que se disocia del DNA.
En realidad la DNA polimerasa III es una proteína oligomérica formada por múltiples subunidades que se coloca en la horquilla de replicación de manera que se encarga simultáneamente de la síntesis en las dos hebras merced a un complejo mecanismo que implica la formación temporal de un lazo en la hebra rezagada a medida que se va formando cada fragmento de Okazak; este lazo desaparece cuando las subunidades del enzima responsables de la síntesis del fragmento se disocian del DNA y se vuelve a formar cuando dichas subunidades se vuelven a incorporar para sintetizar el fragmento siguiente.


Del modo que se ha descrito hasta ahora, cuando las horquillas de replicación en su avance recorran todo el cromosoma bacteriano se habrá completado la replicación en las hebras conductoras mientras que las hebras rezagadas habrán quedado sembradas de fragmentos de Okazaki cada uno de ellos con su correspondiente cebador de RNA.
Las tareas pendientes del equipo enzimático de la replicación consistirán ahora en eliminar los cebadores y sellar la unión de los sucesivos fragmentos de Okazaki. La eliminación de los cebadores es llevada a cabo por la DNA polimerasa I, enzima que, merced a la actividad exonucleasa 5’ à 3’ que lleva incorporada, puede alargar un fragmento dado y al tiempo ir degradando el cebador del fragmento precedente escindiendo uno a uno los ribonucleótidos que lo forman.
Este proceso, conocido como corrimiento de mella, sustituye el cebador por un tramo de ADN recién sintetizado de manera que la hebra rezagada consiste ahora en una sucesión de fragmentos de Okazaki formados exclusivamente por desoxirribonucleótido. Así se eliminan todos los cebadores incluidos los de ambas hebras conductoras, que se ven afectados por la prolongación del primer fragmento de Okazaki sintetizado en las respectivas hebras rezagadas.
Para completar la replicación sólo falta unir estos fragmentos y de ello se encarga un enzima específico, la DNA ligasa, que sella las uniones pendientes entre el último nucleótido de cada fragmento y el primero del siguiente, consumiendo para ello energía de la hidrólisis del ATP. Es conveniente resaltar que, si bien se han descrito como fenómenos separados en el tiempo para facilitar su comprensión, la eliminación de los cebadores y el sellado de los fragmentos de Okazaki discurren en realidad en paralelo con el avance de las horquillas de replicación.


Este proceso se repite cuantas veces sea necesario, a medida que el equipo de helicasas, girasa y topoisomerasava abriendo la doble hélice, hasta completar la replicación del cromosoma. Un fragmento de Okazaki es colocado en la hebra rezagada en cada ciclo mientras que la hebra conductora se sintetiza sin interrupción; cada fragmento finaliza cuando la DNA polimerasa III se encuentra con el cebador del fragmento precedente, momento en que se disocia del DNA.
En realidad la DNA polimerasa III es una proteína oligomérica formada por múltiples subunidades que se coloca en la horquilla de replicación de manera que se encarga simultáneamente de la síntesis en las dos hebras merced a un complejo mecanismo que implica la formación temporal de un lazo en la hebra rezagada a medida que se va formando cada fragmento de Okazak; este lazo desaparece cuando las subunidades del enzima responsables de la síntesis del fragmento se disocian del DNA y se vuelve a formar cuando dichas subunidades se vuelven a incorporar para sintetizar el fragmento siguiente.


Del modo que se ha descrito hasta ahora, cuando las horquillas de replicación en su avance recorran todo el cromosoma bacteriano se habrá completado la replicación en las hebras conductoras mientras que las hebras rezagadas habrán quedado sembradas de fragmentos de Okazaki cada uno de ellos con su correspondiente cebador de RNA.
Las tareas pendientes del equipo enzimático de la replicación consistirán ahora en eliminar los cebadores y sellar la unión de los sucesivos fragmentos de Okazaki. La eliminación de los cebadores es llevada a cabo por la DNA polimerasa I, enzima que, merced a la actividad exonucleasa 5’ à 3’ que lleva incorporada, puede alargar un fragmento dado y al tiempo ir degradando el cebador del fragmento precedente escindiendo uno a uno los ribonucleótidos que lo forman.
Este proceso, conocido como corrimiento de mella, sustituye el cebador por un tramo de ADN recién sintetizado de manera que la hebra rezagada consiste ahora en una sucesión de fragmentos de Okazaki formados exclusivamente por desoxirribonucleótido. Así se eliminan todos los cebadores incluidos los de ambas hebras conductoras, que se ven afectados por la prolongación del primer fragmento de Okazaki sintetizado en las respectivas hebras rezagadas.
Para completar la replicación sólo falta unir estos fragmentos y de ello se encarga un enzima específico, la DNA ligasa, que sella las uniones pendientes entre el último nucleótido de cada fragmento y el primero del siguiente, consumiendo para ello energía de la hidrólisis del ATP. Es conveniente resaltar que, si bien se han descrito como fenómenos separados en el tiempo para facilitar su comprensión, la eliminación de los cebadores y el sellado de los fragmentos de Okazaki discurren en realidad en paralelo con el avance de las horquillas de replicación.
16.3. Fase de terminación
Puesto que la replicación del cromosoma bacteriano comienza en un único origen y es bidireccional, las dos horquillas de replicación avanzando a velocidad constante se encontrarán en el lugar del cromosoma diametralmente opuesto al origen (oriC). Allí, las hebras conductoras de una y otra horquilla de replicación se detendrán al encontrarse con el cebador del último fragmento de Okazaki sintetizado en la hebra rezagada respectiva, que seguidamente será eliminado por la DNA polimerasa I y la mella resultante sellada por la DNA ligasa.
No obstante lo dicho, se ha podido comprobar que el lugar de terminación de la replicación está señalizado por una secuencia específica de nucleótidos (el lugar Ter) a la que se fija una proteína específica que bloquea el avance de las helicasas cuando llegan a ella. Cuando se bloquea el avance de una de las horquillas en un lugar alejado del de terminación la otra horquilla se detiene al llegar a éste de todas maneras.
17. REPLICACIÓN DEL ADN EN EUCARIOTAS
La descripción hasta aquí realizada del proceso replicativo en la bacteria E. coli es aplicable en líneas generales a todas las células procariotas y en muchos aspectos también a las células eucariotas. En ellas la replicación también es semiconservativa, secuencial, bidireccional, continua en la hebra conductora y discontinua en la rezagada, y es llevada a cabo por un equipo de enzimas y proteínas no enzimáticas que, si bien se les ha asignado denominaciones diferentes a las de sus análogas bacterianas, actúan de manera muy similar a ellas. La replicación del ADN eucariota, sin embargo, presenta una serie de particularidades que es conveniente poner de manifiesto:
17. REPLICACIÓN DEL ADN EN EUCARIOTAS
La descripción hasta aquí realizada del proceso replicativo en la bacteria E. coli es aplicable en líneas generales a todas las células procariotas y en muchos aspectos también a las células eucariotas. En ellas la replicación también es semiconservativa, secuencial, bidireccional, continua en la hebra conductora y discontinua en la rezagada, y es llevada a cabo por un equipo de enzimas y proteínas no enzimáticas que, si bien se les ha asignado denominaciones diferentes a las de sus análogas bacterianas, actúan de manera muy similar a ellas. La replicación del ADN eucariota, sin embargo, presenta una serie de particularidades que es conveniente poner de manifiesto:
- Los cromosomas eucariotas presentan múltiples orígenes de replicación que se alternan con las correspondientes secuencias de terminación, de manera que en el proceso replicativo varias burbujas de replicación están activas simultáneamente y van creciendo en ambos sentidos hasta que se funden con sus vecinas. Cada unidad replicativa, incluyendo su origen de replicación y delimitada por las correspondientes secuencias de terminación, recibe el nombre de replicón.

- Las ADN polimerasas eucariotas son sensiblemente más lentas que las bacterianas (algunas decenas de nucleótidos por segundo frente a unos 1.000 nucleótidos por segundo). Tal lentitud se compensa con la existencia de orígenes múltiples de replicación.
- Los fragmentos de Okazaki son más cortos que los de las células procariotas.
- La eliminación de los cebadores es realizada por una exonucleasa 5’ à 3’ que es independiente de la polimerasa. No existe un enzima análogo a la ADN polimerasa I de E. coli. La sustitución de los cebadores por ADN, una vez eliminados éstos, es realizada por las mismas ADN polimerasas responsables de la síntesis de los fragmentos.

Mención aparte merece el problema del final de la replicación en los extremos de los cromosomas eucariotas denominados telómeros. Este problema no se da en las células procariotas por presentar éstas cromosomas circulares y por lo tanto carentes de extremos.
En los telómeros la replicación de la hebra conductora finaliza sin complicaciones ya que las DNA polimerasas finalizan la síntesis en el lugar exacto en el que finaliza el molde. Por el contrario, en la hebra rezagada el cebador del último fragmento de Okazaki es colocado exactamente en el extremo 5’, emparejado con la cadena molde; cuando la exonucleasa 5’ à 3’ elimina este cebador ninguna DNA polimerasa podrá rellenar el hueco que deja. De este modo, la replicación queda inconclusa en el extremo 5’ y cuando en el siguiente ciclo replicativo la cadena así acortada sirva de molde para la síntesis de su complementaria se habrá perdido definitivamente un tramo de la cadena polinucleotídica.
El acortamiento de los telómeros en cada ciclo de división celular a causa del fenómeno descrito puede suponer la pérdida de información genética importante y con ello el deterioro y la muerte de la célula. Para paliar este grave problema los organismos eucariontes se han dotado de un enzima, la telomerasa, que tiene la función de reparar los telómeros en cada ciclo de división celular evitando así su acortamiento.
El acortamiento de los telómeros en cada ciclo de división celular a causa del fenómeno descrito puede suponer la pérdida de información genética importante y con ello el deterioro y la muerte de la célula. Para paliar este grave problema los organismos eucariontes se han dotado de un enzima, la telomerasa, que tiene la función de reparar los telómeros en cada ciclo de división celular evitando así su acortamiento.
La telomerasa es un enzima con actividad de DNA polimerasa que lleva incorporado como grupo prostético un oligonucleótido de RNA que sirve de molde para sintetizar una cadena complementaria de DNA. Los telómeros poseen unas secuencias repetitivas de DNA parcialmente complementarias con el molde de RNA interno de la telomerasa. Cuando estas secuencias se aparean, dicho molde interno es utilizado para alargar el extremo 3’ sobresaliente del telómero. Se producen varios ciclos de alargamiento y a continuación la maquinaria replicativa ordinaria coloca un fragmento de Okazaki adicional en el extremo 5’ defectuoso, de manera que, aunque posteriormente se eliminará el cebador de este fragmento, el tramo de DNA así sintetizado compensa el acortamiento del telómero.

Se ha especulado mucho con la idea de que el acortamiento de los telómeros en las células somáticas pueda estar relacionado con los procesos de envejecimiento general del organismo y de que una potenciación de la actividad de la telomerasa en estas células pudiera resultar útil para retrasarlo. Existe un grave inconveniente: la actividad de la telomerasa es particularmente alta en la mayoría de las células tumorales. En realidad, la desactivación de este enzima en las células somáticas podría funcionar como un eficaz mecanismo de prevención del cáncer, ya que el acortamiento de los telómeros actuaría como una especie de reloj biológico que conduciría a la muerte celular antes de que una célula pueda acumular las mutaciones necesarias para convertirse en tumoral. Por el contrario, las células portadoras de una mutación que reactive la telomerasa serían candidatas a convertirse en cancerígenas. El bloqueo selectivo de la actividad de la telomerasa en estas células podría constituir una vía para el tratamiento de esta enfermedad.
| 18. IDEAS FUNDAMENTALES |
La célula es la unidad de vida.
Las células sin núcleo son procariotas.
Las células con núcleo son eucariotas animales o vegetales.
El núcleo es la parte de la célula que contiene la información genética.
Las células con núcleo son eucariotas animales o vegetales.
El núcleo es la parte de la célula que contiene la información genética.
La envoltura nuclear es doble membrana con poros que permiten el intercambio de sustancias con el citoplasma.
El nucléolo es una esfera que flota en el nucleoplasma que tiene como función la síntesis de ARN que forma los ribosomas.
El ADN del núcleo se asocia a proteínas histónicas y forma la cromatina que es el estado desempaquetado de los cromosomas.
El nucléolo es una esfera que flota en el nucleoplasma que tiene como función la síntesis de ARN que forma los ribosomas.
El ADN del núcleo se asocia a proteínas histónicas y forma la cromatina que es el estado desempaquetado de los cromosomas.
Los cromosomas son las estructuras celulares que contienen y mantienen, en la reproducción celular, la información genética.
Un GEN es un fragmento de DNA que posee la información necesaria para desarrollar un carácter.
La RECOMBINACIÓN GENÉTICA es un mecanismo por el cual dos cromosomas homólogos intercambian entre sí fragmentos de DNA equivalentes, es decir, intercambian los mismos genes o trozos de genes, aunque pueden tener alelos diferentes. La REPLICACIÓN del DNA consiste en hacer una copia idéntica de la doble hélice, según el modelo semiconservativo.
La RECOMBINACIÓN GENÉTICA es un mecanismo por el cual dos cromosomas homólogos intercambian entre sí fragmentos de DNA equivalentes, es decir, intercambian los mismos genes o trozos de genes, aunque pueden tener alelos diferentes. La REPLICACIÓN del DNA consiste en hacer una copia idéntica de la doble hélice, según el modelo semiconservativo.
19. REPASO
23. OTRAS PRESENTACIONES
24. CUESTIONES
Célula 1
Célula 2
Célula 3
Célula 4
Dibujos células 2
Preguntas selectividad importantes
Preguntas selectividad célula 1
Preguntas selectividad célula 2
Preguntas selectividad célula 3
Preguntas selectividad célula 4
Dibujos de célula animal y vegetal
Dibujos para completar de células
Núcleo
23. OTRAS PRESENTACIONES
24. CUESTIONES
Célula 1
Célula 2
Célula 3
Célula 4
Dibujos células 2
Preguntas selectividad importantes
Preguntas selectividad célula 1
Preguntas selectividad célula 2
Preguntas selectividad célula 3
Preguntas selectividad célula 4
Dibujos de célula animal y vegetal
Dibujos para completar de células
Núcleo